Puce de microfluidie pour l’observation de racines de plantules (Y LERENDU, GlycoMEV/CMAbio3 et ( S. BEGREM ABTE/CMAbio3)

Le Centre de microscopie appliquée à la biologie (CMAbio3) conseille, accompagne et forme ses utilisateurs à l’utilisation de différents types de microscopes photoniques et électroniques, d’équipements de préparation d’échantillons et de microfluidique.
Spécialisée dans la préparation des échantillons pour la microscopie électronique en biologie, la plateforme dispose aussi de quelques équipements pour préparer les échantillons pour la microscopie optique.
Le service dispose d’une chaîne de fabrication de puces microfluidique par photolithographie et d’une fraiseuse 5 axes pour fabriquer des systèmes de cultures plus grand. Un système de gestion microfluidique est aussi disponible.
Enfin, la plateforme propose un service de traitement et d’analyse des images d’échantillons biologiques obtenues à partir de nos systèmes ou d’autres capteurs. Ils sont réalisés à l’aide de logiciels libres et des applications plus spécifiques peuvent être développées en interne. Le personnel du CMAbio3 effectue des prestations, du conseil, forme les étudiants, techniciens, chercheurs de l’université ou les utilisateurs externes à l’établissement, à toutes les techniques de préparation, d’observation des échantillons et de traitement/analyse des images.
Les activités sont organisées sur trois plateaux techniques :
CMAbio
Le plateau de microscopie (CMAbio) est équipé pour l’observation d’échantillons aux différentes échelles tissulaires, cellulaires et ultrastructurales par microscopie photonique et électronique. Ce plateau technique peut prendre en charge toutes les étapes du traitement d’échantillons biologiques et leur observation en microscopie électronique à transmission (MET). L’ingénieur a aussi un accès aux MEB, MEB-EDS, LVMEB du CRISMAT (ENSICAEN). Le service dispose d’un vibratome, d’une cytocentrifugeuse d’un appareil à point critique,d’un systéme de micro-injection, d’un ultramicrotome, d’un cryoultramicrotome, d’un système de cryobusbtitution et d’un système de cryotransfert pour MEB. Nous disposons aussi d’équipements pour des diverses modalités d’observation en macroscopie, microscopie lumière blanche/fluoresence et microscopie confocale à balayage laser. Le personnel affecté au plateau accompagne les chercheurs dans leurs travaux en leur transmettant leur savoir-faire et accueille des étudiants au cours de formations courtes pour leur faire découvrir ces technologies.
AIM
Le plateau d’analyse d’images multimodales (AIM) accompagne les travaux de recherche des équipes institutionnelles et industrielles, pour la mise au point de procédures de traitement et d’analyses d’images d’échantillons biologiques. Il a également une mission de formation et de conseil sur les techniques d’acquisition, de traitement et d’analyse d’images pour les chercheurs mais aussi pour les étudiants de l’université.
MicroFluDesign
Le plateau MicroFluDesign qui est un plateau dédié à la conception et la production de puces/systèmes microfluidiques dédiés à la biologie. Les équipements permettent le développement de compétences synergiques et originales en microfluidique, microscopie et analyses de micro-environnements. Ces approches pourront être couplées à la microscopie et l’analyse chimique et moléculaire de micro-environnements complexes pour la caractérisation du vivant et de la biodiversité. Des approches continue (micro canaux) ou discrète (de gouttes dite microfluidique digitale) peuvent permettre d’effectuer des opérations variées en parallèle sur un grand nombre d’échantillons basées notamment sur du tri cellulaire (à des fins de diagnostic ou biotechnologique). Ces dispositifs d’analyse microfluidique sont fabriqués le plus souvent avec les outils de la microélectronique, et peuvent par exemple être des « organes sur puces » ou des « plantes sur puces ». L’ensemble du personnel du service va acquérir ces techniques pour les mettre au service de la communauté. Venez avec vos questions et vos échantillons.
Préparation d’échantillons

Appareil à contournement du point critique CPD030 LEICA 
Appareil de glow discharge ELMO CORDOUAN 
Cryo-ultramicrotome, RMC Powertome coupe ultra-fine d’échantillon congelé 
Ultramicrotome, coupe Semi-fines et Ultra-Fines Leica Ultracut R 
Système de cryosubstitution 7500 RMC 
Cytosmin 4 Thermo 
Vibratome
Microscopie

Microscope inversé axio observer zeiss 
Microscope électronique à balayage LV EDS JEOL 7200 du CRISMAT 
Microscope confocal FV1000 olympus 
Microscope olympus BX53 
Macroscope Axio zoom Zeiss 
Macroscope Axio zoom Zeiss 
Microscope electronique à transmission 10 11JEOl 
Miccroscope électronique à balayage EDS ZEISS-SUPRA55-1du CRISMAT avec cryotransfert Quorurm

Puces en PMMA (plexiglass) ou en PDMS (silicone) avec lamelle de microscopie 
Puces en PMMA (plexiglass) ou en PDMS (silicone) avec lamelle de microscopie 
Puces en PMMA (plexiglass) ou en PDMS (silicone) avec lamelle de microscopie 
puce thermostatée au microscope 
puce au microscope 
puce thermostatée au microscope 
puce thermostatée au microscope 
Polymérisation par écriture laser 
Polymérisation par écriture laser 

Microfraisage de puce sur PMMA 
Microfraisage de puce sur PMMA 
Microfraisage de puce sur PMMA 
Microfraisage de puce sur PMMA 
Microfraisage de puce sur PMMA 
Macroscope 
Imprimante 3D 
Système de gestion des flux Fluigent 
Spin coater pour le dépôt homogène de résine de quelques dizaine, centaine de nm d'épaisseur 
Système de nettoyage au plasma 
fraiseuse 5 axes Pocket NC 
Systéme d'eccriture laser Dilsae 125 KLOE
Le personnel pourra vous accompagner sous la forme de prestation, de formation puis de travaux autonomes :

Puces de microfluidie
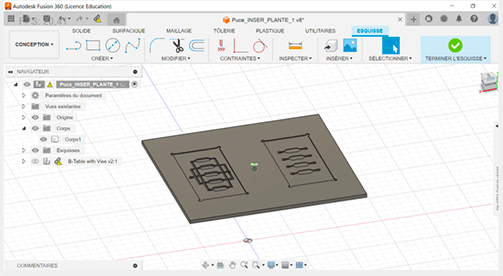
Conception 3D avec le logiciel Fusion 360 (AUTODESK)

Microscopie grand champs : silique de Colza
Silique de Colza (image mosaïque au macroscope Axio zoom ZEISS) (EVA JC AVICE; BRUNEL-MUGUET S CMAbio3)

Plantule observée au macroscope
Plantule d’Arabidopsis thaliana observée au macroscope Axio Zoom Zeiss (GlycoMEV CMAbio3, Y. LERENDU)
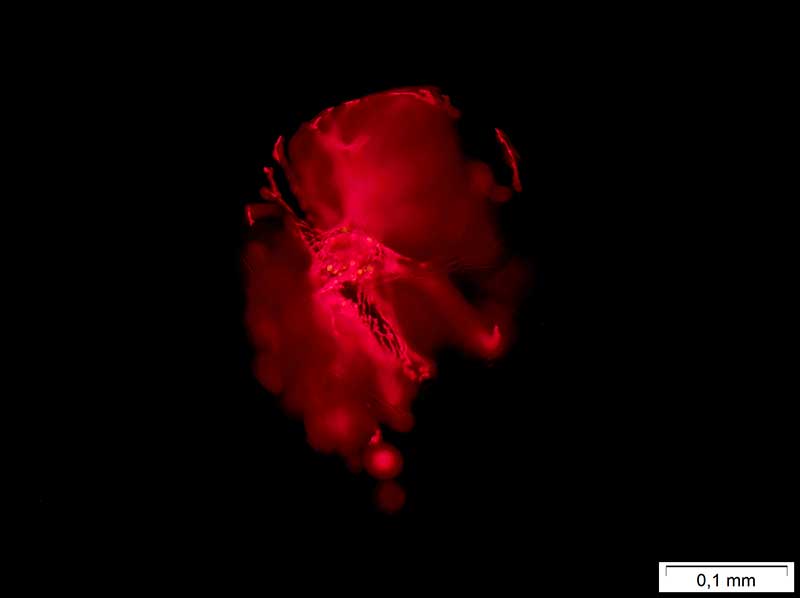
Microscopie : organisme bioluminescent (Pyrocystis sp.)
Image acquise en microscopie optique (Olympus) de Pyrocystis sp.(CMAbio3)
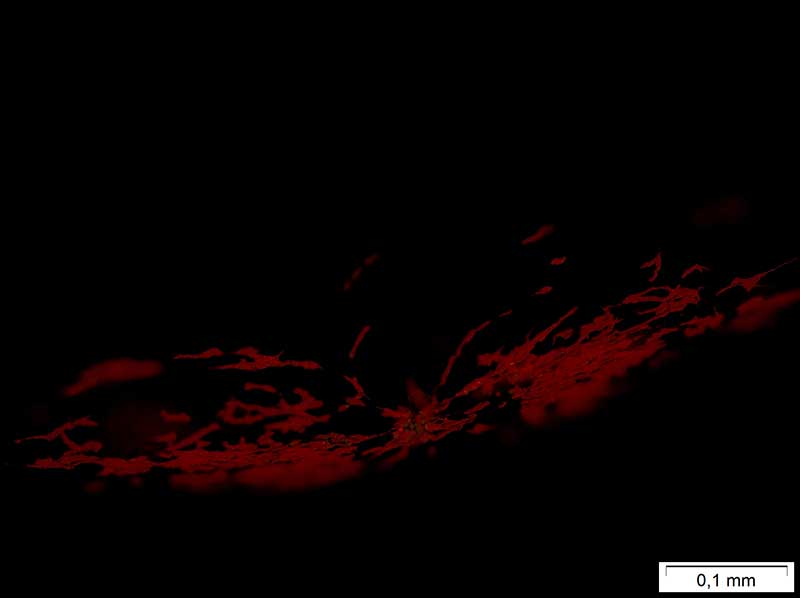
Pyrocystis sp.
Image acquise en microscopie de Pyrocystis sp.(CMAbio3)
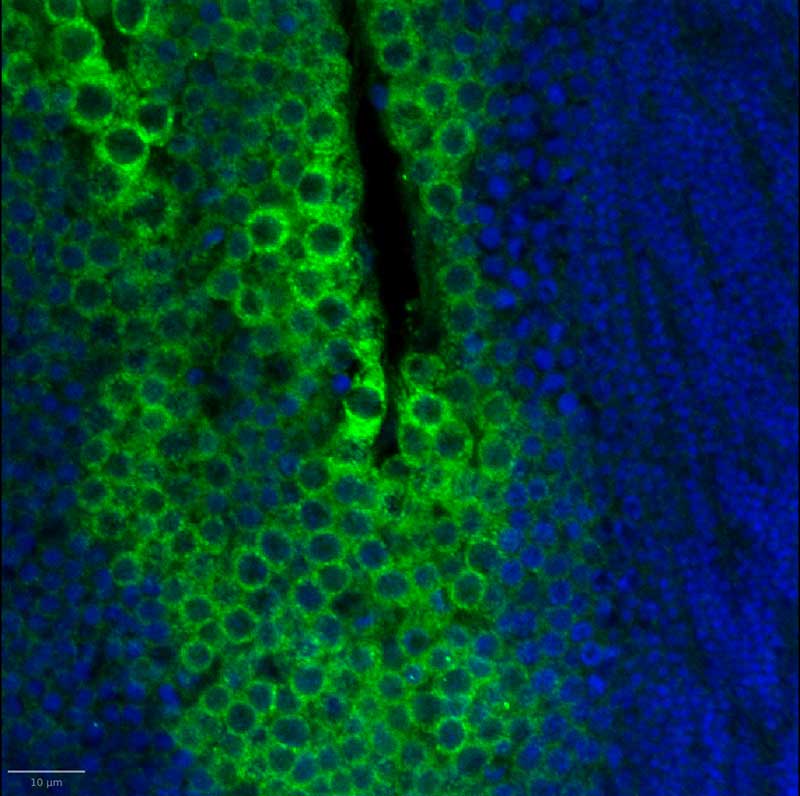
Coupe paraffine d’huître, immunomarquage
Coupe paraffine d’huitre, tubule gonadique (Crassostrea gigas), marquage DAPI/VASA, image microscopie confocal Olympus FV1000 (echelle 20µm). CMAbio3; BOREA CHERIF-FEILDEL M. KELLNER K., BERTHELIN C.)
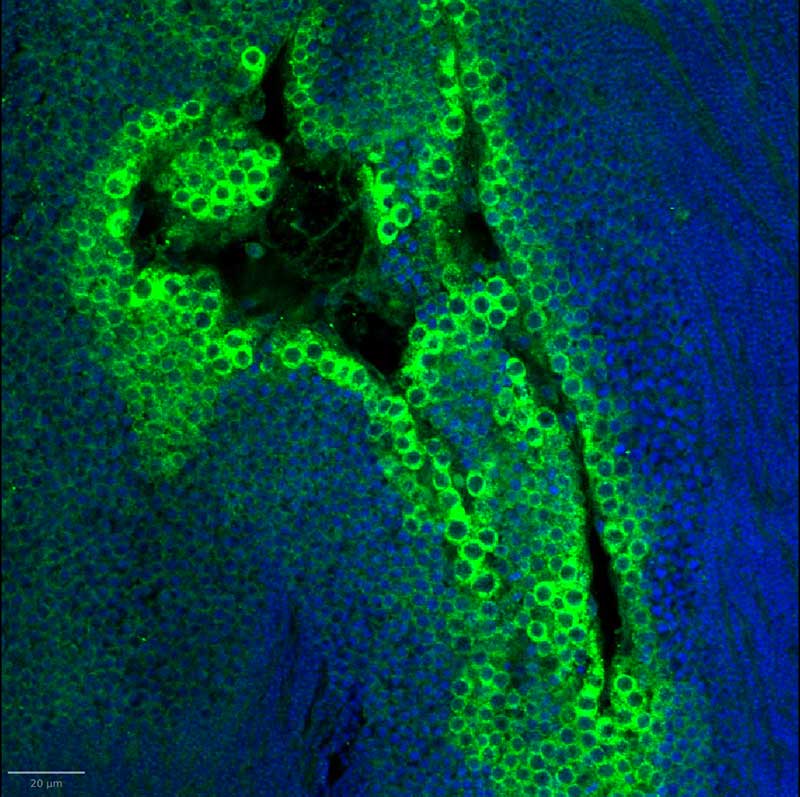
Immunomarquage DAPI/VASA sur coupe paraffine d’huître
Coupe paraffine d’huitre, tubule gonadique (Crassostrea gigas ), marquage DAPI/VASA, image microscopie confocal Olympus FV1000 (echelle 20µm). CMAbio3; BOREA CHERIF-FEILDEL M. KELLNER K., BERTHELIN C.)
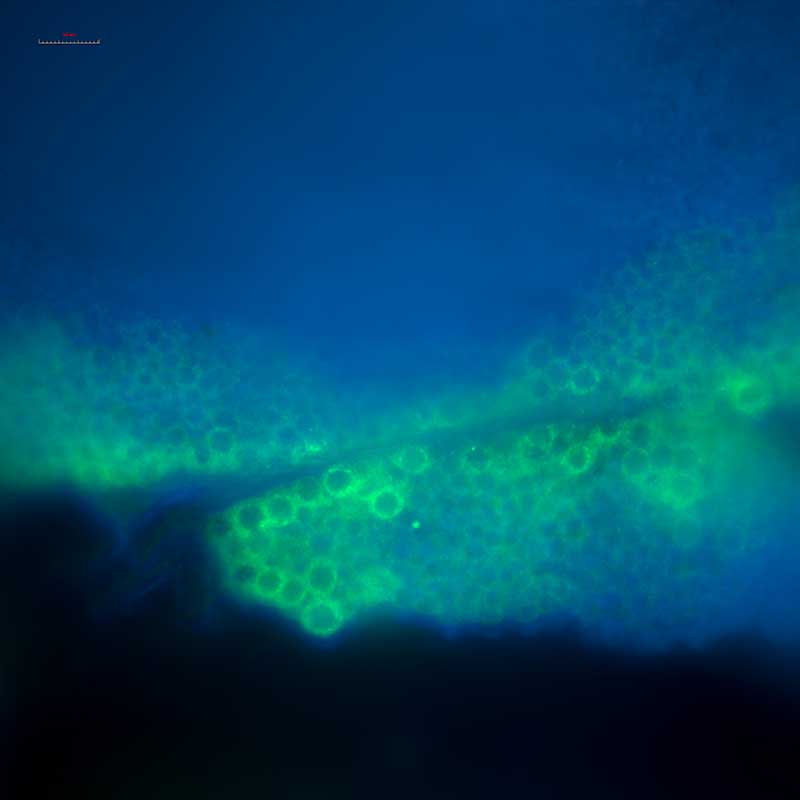
Immunomarquage VASA /DAPI sur coupe vibratome d’huître
Coupe vibratome d’huitre, tubule gonadique (Crassostrea gigas ), marquage DAPI/VASA (echelle 10µm). CMAbio3; BOREA CHERIF-FEILDEL M. KELLNER K., BERTHELIN C.)
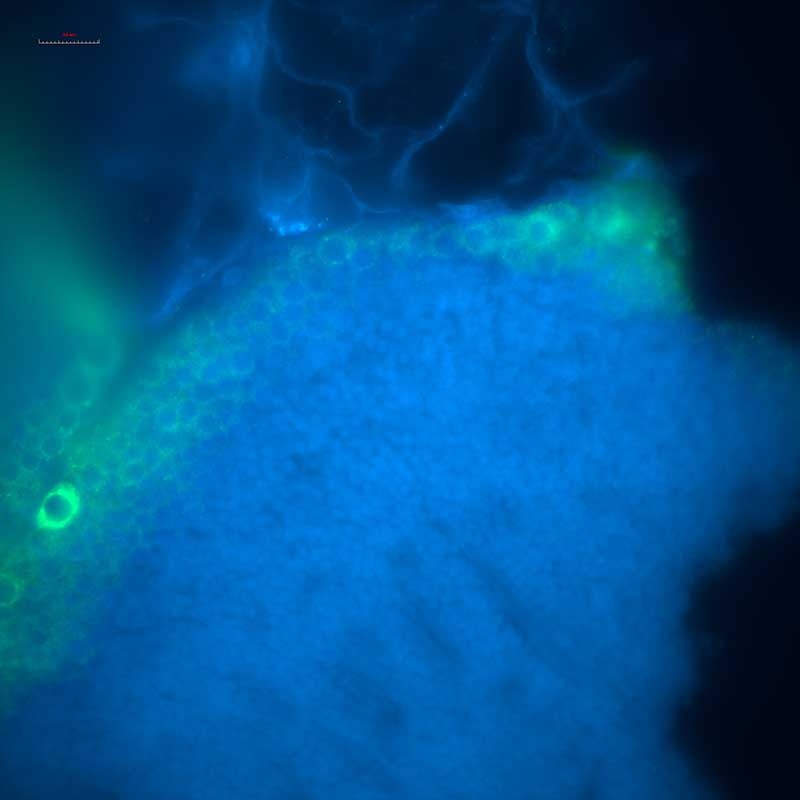
Immunomarquage VASA /DAPI sur coupe vibratome d’huître
Coupe vibratome d’huitre, tubule gonadique (Crassostrea gigas ), marquage DAPI/VASA, image acquise au microscope confocal Olympus FV100 (echelle 10µm). CMAbio3;BOREA CHERIF-FEILDEL M. KELLNER K., BERTHELIN C.)
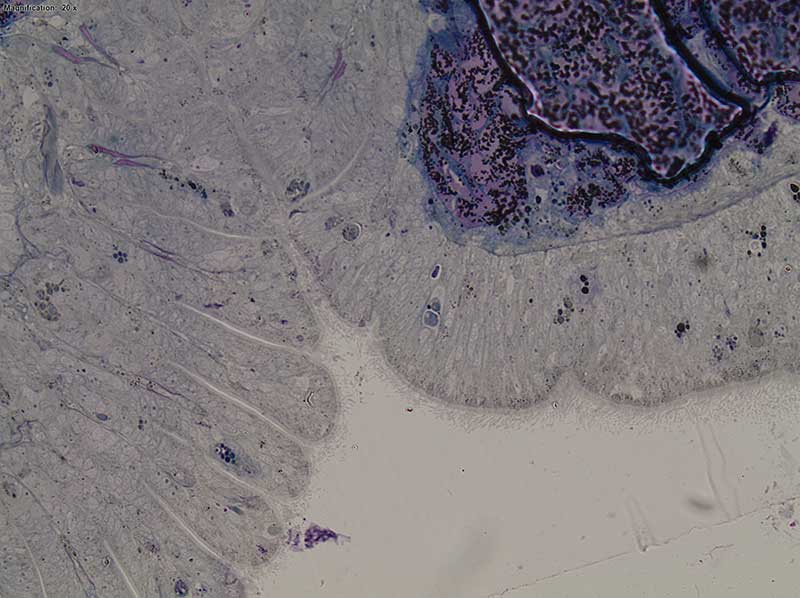
Coupe semi fine de branchie d’huitre
Coupe semi fine de branchie d’huître (RIN SHEMAH, BOREA-CMAbio3 (KELLNER K., BERTHELIN C., GOUX D., RIVIERE G., ELIE N., ARREDONDO R.C. et H. AUGER)

Coupe feuille Colza
Coupe vibratome de Colza (Cylia HADDAD, P. ETIENNE, P. LAINE EVA CMAbio3)

Fraisage PMMA
Fraisage PMMA (LERENDU Y) (GLYCOMEV/CMABIO3) photo MH LABAT

Flux laminaires
Puces microfluidique, flux presque laminaires (LERENDU Y CMAbio3)

Puces de microfluidie pour plantules
Puce de microfluidie pour l’observation de racines de plantules (Y LERENDU, GlycoMEV/CMAbio3)

Anayse d’images (caméras automatiques)
Détection automatique de règles : identification, et mesure de la hauteur de la règle sur des séries de photos jour et nuit (unité IDEES – Romain REULIER)

Dénombrement automatique de graines
Dénombrement automatique de graines : Détection et dénombrement de graines (unité EVA – Julien Besnard)
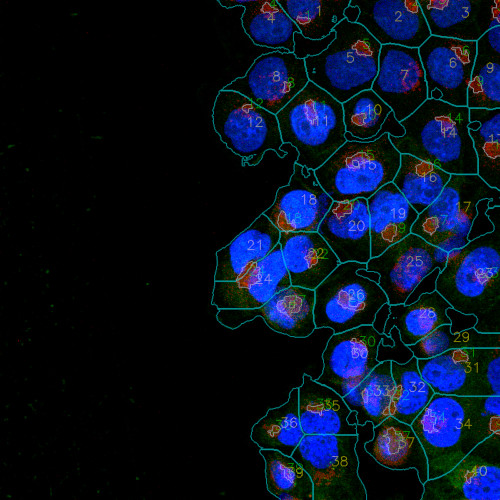
Détection de territoires cellulaires et de marqueurs
Détection de territoires cellulaires et de marqueurs : Culture cellulaire acquise au microscope confocal sur trois canaux et détection des cellules avec les noyaux, limites des cellules et marqueurs internes (unité ISCTC – Tiphaine Biojout)

Détection de chromatophores de peau de seiche
Détection chromatophores sur peau de seiche : Extrait d’une séquence d’images de peau de seiche sur laquelle les chromatophores sont individualisés et leurs surfaces évaluées (Projet Inter-Reg REPOL – unité Ethos – Anaid Gouveneaux)
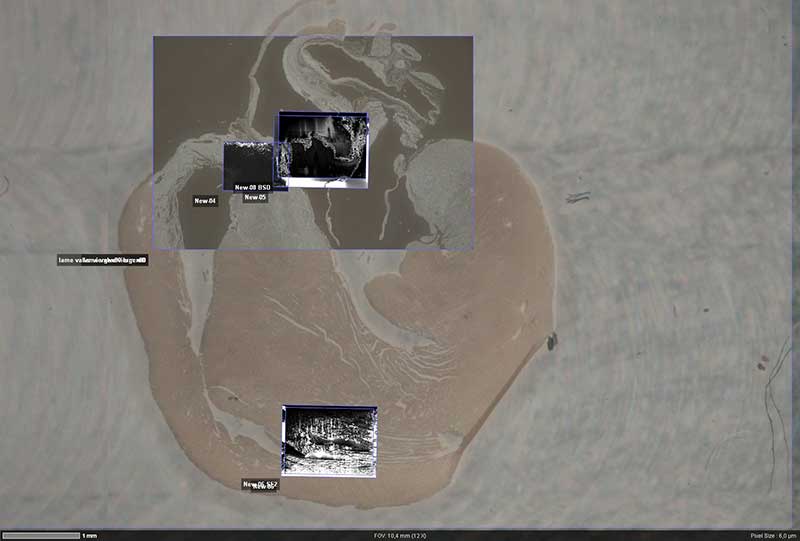
Microscopie corrélative
Coupe paraffine de coeur de souris observée au macroscope avio zoom Zeiss puis au MEB zeiss supra 55 (Collaboration Virtual His; CMAbio3/CRISMAT/PSIR) logiciel Zeiss Zen
Fusion 360, ZEN, ZEN Connect, Olympus CellSens, Digital Micrograph, Qupath, ImageJ, FIJI, Ilastik, OMERO
Contactez nous en fonction de vos besoin et/ou utilisez ce questionnaire
Formulaire de demande d’accès du CMAbio3
Formulaire de satisfaction anonyme si vous le souhaitez
Contactez nous pour que nous vous aidions à préparer vos séances/échantillons
À consulter
Voici un document qui résume quelques conseils de préparation pour la microscopie électronique
L’ensemble des plateformes est regroupé sur le campus 1 de l’université.


nicolas.elie@unicaen.fr
Tel : 02 31 56 58 81
Tel : 02 31 56 58 13


