Détection automatique de règles : identification, et mesure de la hauteur de la règle sur des séries de photos jour et nuit (unité IDEES – Romain REULIER)

Le plateau d’analyse d’images multimodales (AIM) du CMAbio3 accompagne les travaux de recherche des équipes institutionnelles et industrielles, pour la mise au point de procédures de traitement et d’analyses d’images d’échantillons biologiques. Il a également une mission de formation et de conseil sur les techniques d’acquisition, de traitement et d’analyse d’images pour les chercheurs mais aussi pour les étudiants de l’université.
Serveurs / Logiciel
- ZEN,
- ZEN Connect,
- Olympus CellSens,
- Digital Micrograph,
- Qupath,
- ImageJ,
- FIJI,
- Ilastik,
- OMERO
Exemples
Dénombrement automatique de graines
Dénombrement automatique de graines : Détection et dénombrement de graines (unité EVA – Julien Besnard)
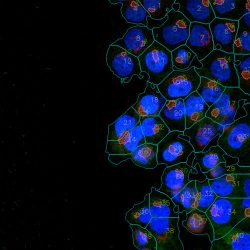
Détection de territoires cellulaires et de marqueurs
Détection de territoires cellulaires et de marqueurs : Culture cellulaire acquise au microscope confocal sur trois canaux et détection des cellules avec les noyaux, limites des cellules et marqueurs internes (unité ISCTC – Tiphaine Biojout)
Détection de chromatophores de peau de seiche
Détection chromatophores sur peau de seiche : Extrait d’une séquence d’images de peau de seiche sur laquelle les chromatophores sont individualisés et leurs surfaces évaluées (Projet Inter-Reg REPOL – unité Ethos – Anaid Gouveneaux)